Los métodos de bioinformática estadística se pueden aplicar a cualquier conjunto de datos biológicos, no solo a datos derivados de NGS.
Además de las fuentes de datos individuales, la integración de múltiples tipos de datos permite el acceso a conocimientos que serían imposibles de recopilar a partir de un solo tipo de conjunto de datos.
MÁS INFORMACIÓN SOBRE EL ANÁLISIS QUE OFRECEMOS
El análisis funcional se ha convertido en la primera opción para obtener información sobre la biología subyacente de genes y proteínas expresados diferencialmente, ya que ayuda a interpretar los datos en el contexto de procesos biológicos, rutas y redes. Los genes implicados en los mismos procesos biológicos, funciones o localizaciones suelen presentar comportamientos correlacionados en términos de niveles de expresión, intensidad de señales, apariciones de alelos, etc. Por tanto, podemos aplicar pruebas estadísticas para encontrar funciones o vías afectadas. Por lo general, las dos fuentes de información para este tipo de análisis son la base de conocimiento de Ontología Genética (GO) y la base de datos completa de vías metabólicas denominada Enciclopedia de genes y genomas de Kyoto (KEGG). Ofrecemos el siguiente análisis: Dada una lista de genes expresados diferencialmente bajo ciertas condiciones, una ORA probará qué términos o vías de GO están sobre o subrepresentados usando la anotación pública de esos genes. Casos de uso El análisis funcional podría aplicarse a datos de diferentes fuentes: Los microarrays se pueden usar en muchos tipos de experimentos, incluido genotipado, epigenética, perfiles de traducción y perfiles de expresión génica. El perfil de expresión genética es probablemente el uso más común de la tecnología de microarrays. Se pueden utilizar microarrays de uno y dos colores para este tipo de experimentos. El resultado de un análisis de proteoma suele ser una larga lista de proteínas con una puntuación de probabilidad e, idealmente, un valor cuantitativo asociado. Así, al igual que con los conjuntos de genes, algunos de los análisis bioinformáticos de datos de proteómica incluyen anotación funcional y análisis de enriquecimiento, agrupamiento, análisis de redes u otros análisis estadísticos, o análisis de modificaciones postraduccionales. Casos de uso El análisis bioestadístico siempre ha sido un componente clave de los estudios biológicos, y especialmente genómicos. Tenemos una amplia gama de experiencia en diseño experimental y estadístico. Todos nuestros servicios pueden adaptarse para satisfacer las necesidades de tu proyecto: Casos de uso Nuestra instalación tiene experiencia en la realización de una amplia gama de diversos análisis estadísticos: A petición de nuestros usuarios, diseñamos software especializado para resolver problemas biológicos específicos. Tenemos una gran experiencia en la escritura de scripts internos en lenguajes de programación como R, Python, Awk y Perl, así como en el lenguaje de comandos bash.Análisis de enriquecimiento funcional
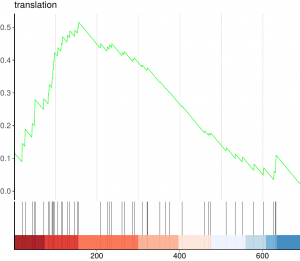 Dado un conjunto de genes definido a priori (por ejemplo genes que codifican productos en una vía metabólica o que comparten la misma categoría GO) y una lista clasificada de genes expresados diferencialmente (por ejemplo ordenados por su expresión), el objetivo de GSEA es determinar si los miembros del conjunto se distribuyen aleatoriamente en la lista o se encuentran principalmente en la parte superior o inferior. La puntuación de enriquecimiento (ES) representa la cantidad en la que los genes del conjunto están sobrerrepresentados en la parte superior o inferior de la lista.
Dado un conjunto de genes definido a priori (por ejemplo genes que codifican productos en una vía metabólica o que comparten la misma categoría GO) y una lista clasificada de genes expresados diferencialmente (por ejemplo ordenados por su expresión), el objetivo de GSEA es determinar si los miembros del conjunto se distribuyen aleatoriamente en la lista o se encuentran principalmente en la parte superior o inferior. La puntuación de enriquecimiento (ES) representa la cantidad en la que los genes del conjunto están sobrerrepresentados en la parte superior o inferior de la lista.
Análisis de microarrays
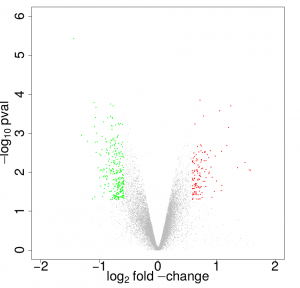
Proteómica
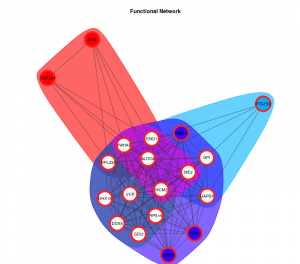
Estadística

Software personalizado